****研究介绍(节选)
当T细胞受体(TCRs)识别抗原递呈细胞上的,与组织相容性复合体分子结合的激动剂,T细胞就会被激活。T细胞的激活很大程度上依赖于质膜上TCRs的时空排列。然而,由于光的衍射极限, T细胞上的TCRs的分子组织尚不明确。本文通过直接随机光学重建显微镜和结构照明显微镜(SIM)观察了淋巴结中纳米尺度和微米尺度的TCRs分布。这种d和SIM方法为体内免疫反应过程中TCRs的多尺度重组提供了**个证据。在未活化T细胞上观察到纳米尺度的质膜结构域,称为蛋白质岛。这些蛋白质岛以微米大小的尺寸在表面区域内富集。在体内,T细胞的激活使TCRs区域收缩,导致蛋白岛的聚集,形成稳定的TCR微簇。
NO.2研究结果(节选)
1.在未活化的淋巴结T细胞上,d可视化纳米级TCR岛
d成像显示,在初始淋巴结驻留的T细胞上存在纳米级的TCR岛(图1A)。用Alexa Fluor 647染料(AF-647)直接偶联β链的单克隆抗体(H57)检测TCRs。同时检测细胞表面和横截面表明在T细胞的质膜上有特异性的染色。基于TIRF的d图像,用于区分TCR岛和单个TCRs。TCRs在5c.c7 细胞的T细胞质膜上呈有序分布(图1B)。将TCR岛定义为间隔小于2.5倍定位精度的TCRs集合(图1B中重叠的圆圈)。大多数蛋白质岛显示出更大的尺寸,不对称的形状(图1 B和C)。
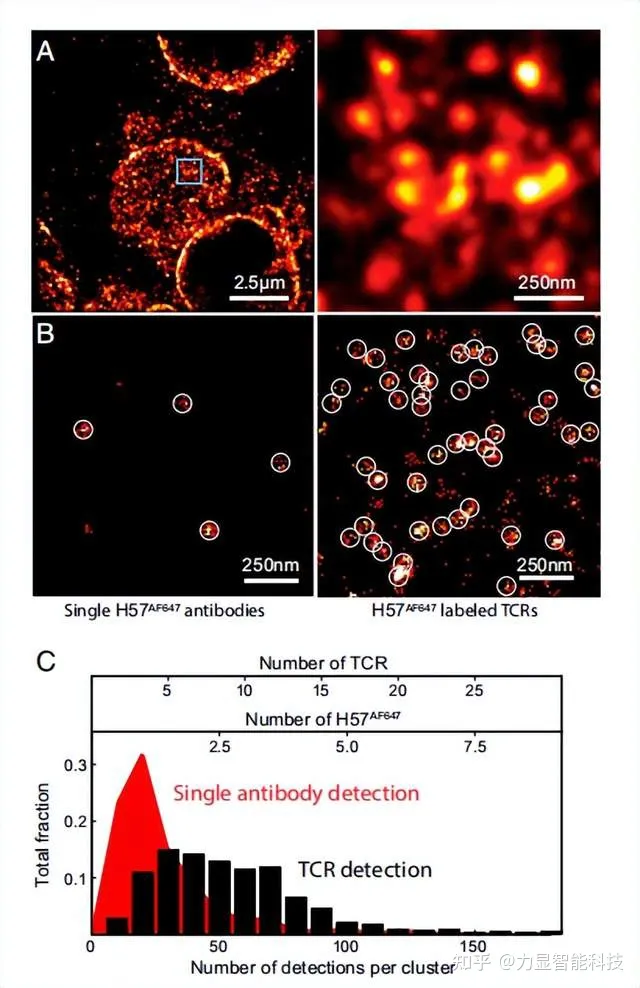
图1 未活化的淋巴结T细胞上TCRs的超分辨率成像
(A)未活化T细胞上TCRs的d图像。(B)H57-AF647单分子的聚类检测(左),使用基于TIRF的d标记有H57-AF647的TCR岛(右)。圆圈突出显示了由单一抗体产生的检测事件。(C)每个单个抗体(红色)和TCR蛋白岛(黑色)检测到的事件数的表征。
2.d表明T细胞激活后形成TCR微簇
与未注射MCC肽的小鼠T细胞相比,注射MCC肽的小鼠T细胞在形状、大小和膜粗糙度上的不均一性增加(图2A),与未活化的T细胞相比,活化的T细胞中的一个TCR子集大小为200-800 nm(图2B)。这些结构类似于上述的微团簇。超分辨率图像中的局部荧光密度峰表明蛋白质岛之间的分离减少(图2C)。
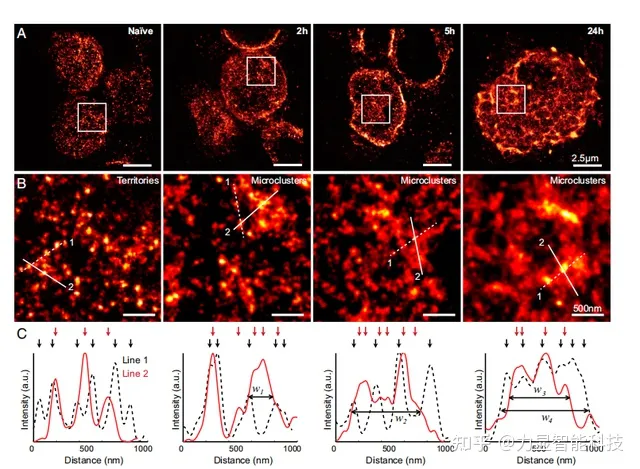
图2 体内T细胞活化前后淋巴结中TCRs的d成像
(A)未注射MCC肽的小鼠和注射MCC肽后2、5和24小时的小鼠T细胞的超分辨图像。(B)图A白色框中的放大图像(C)B中线条的强度分布。箭头标记代表蛋白质岛的局部强度峰值。相邻蛋白质岛之间的距离减小导致强度峰的重叠和结构的加宽(w1=240,w2=507,w3=533,w4=753nm)。
3.淋巴结内T细胞中的TCR岛和微簇的SIM成像
使用SIM确认d的发现。在未活化的的T细胞上,SIM检测到分离的TCR岛,尺寸为100-300nm(图3A-C)。T细胞在体内活化后,TCR分布有明显变化,组装间分离距离为200 ~ 500 nm(图3D-F)。
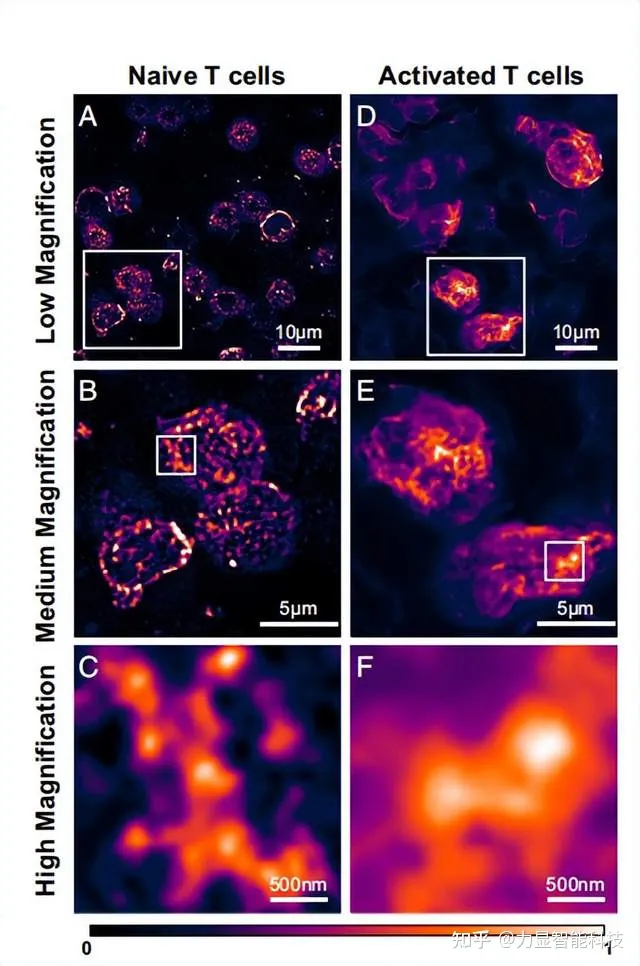
图3 体内T细胞激活前后淋巴结TCR的SIM成像
(A)未活化T细胞的低倍放大图。(B)图A白色方框的放大图,显示蛋白质岛。(C)图B中白色方框的放大图,显示其中的TCR区域和蛋白质岛。(D)肽注射后5小时体内活化T细胞的低倍放大图。(E)D中白色方框的放大图,显示质膜上TCRs密度高的区域。(F)E中白色方框的放大图,显示一个TCR微簇。
NO.3研究总结
总之,本文研究了淋巴结中T细胞在体内激活前后的TCR的组织重排,实现了亚衍射分辨率,并分辨了未活化T细胞上的TCRs蛋白岛和区域。在注射了激动剂多肽的小鼠中,观察到了微簇的形成。本研究为体内TCRs的空间组织结构提供了多条证据。
参考文献
References
Hu YS, Cang H, Lillemeier BF. Superresolution imaging reveals nanometer- and micrometer-scale spatial distributions of T-cell receptors in lymph nodes. Proc Natl Acad Sci U S A. 2016 Jun 28;113(26):7201-6. doi: 10.1073/pnas.1512331113. Epub 2016 Jun 14. PMID: 27303041; PMCID: PMC4932922.